Rでk-means法とその拡張2 x-means編
あっ, どーも僕です。
注意!!本エントリーの記述はRソースを含めて全面的に書き直しています!!(なぜかこのエントリーはアクセス数が多いので(汗))@2014/9/11
Python版はこちらから.
クラスター数が自動で決まるkmeans法
前回はk-means法の実装を示しました。
kmeans法は近い者同士を集めるという直感的に理解しやすいシンプルなクラスタリングですが、そのシンプルさゆえにいくつか欠点(?)があります。
その一つはクラスター数の設定です。
kmeans法ではクラスター数をパラメータとして与えます。...しかし、どうやってクラスター数を設定するればよいのでしょう。...わかりません。職人芸でしょうか。
素人にはクラスター数は決められないでしょうか?
考えるめんどくさいのでアルゴリズムに決めてもらいたいです。
そんなクラスター数をアルゴリズムに決めてもらう都合の良いkmeans法があるのでしょうか?
あります。
それがxmeans法です。
xmeans法では情報量基準で改善される限りクラスター数2のkmeans法を再帰的に適用していくものです。
ここではxmeans法のうち石岡によるものをRで実装したコードを示します(コードの内容は微妙に違いますが結果は同じです)。
石岡によるxmeansはシンプルなxmeans法によるクラスタリング結果を、情報量基準で改良されるならば結果をまとめていこうというものです。詳しくは論文を参照してください。
ちなみに、書き直す前のRコードと異なるのはkmeans法におけるkmeans++による初期値の設定、xmeans法における再帰の活用、リファレンスクラスの利用の三点です。コードは3つに分かれていますのでご注意を。なにかご質問がありましたら気軽にどうぞ。
# *********************************************************** # s-kmeans.r # *********************************************************** s_kmeans <- setRefClass ( Class = "s_kmeans", methods = list ( initCluster_random = function (dat, NUM_CLUSTER) { index <- 1:nrow (dat) center_index <- sample (index, NUM_CLUSTER, replace = FALSE) cur_center <- list () for (i in seq_len (NUM_CLUSTER)) cur_center[[i]] <- unlist (dat[center_index[i],]) dist <- sapply (seq_len (NUM_CLUSTER), function (k) { center_ <- cur_center[[k]] rowSums ((sweep (dat, 2, center_, "-")) **2) }) cur_clusters <- max.col (-dist) return (list (cur_center = cur_center, cur_clusters = cur_clusters)) }, initCluster_kmeanspp = function (dat, NUM_CLUSTER) { index <- 1:nrow (dat) center_index <- sample (index, 1) center_ <- unlist (dat[center_index, ]) min_dist <- rowSums ((sweep (dat, 2, center_, "-")) ** 2) while (length (center_index) < NUM_CLUSTER) { prob <- min_dist[-center_index] / sum (min_dist[-center_index]) new_center <- sample (index[-center_index], 1, prob = prob) center_ <- unlist (dat[new_center, ]) new_dist <- rowSums ((sweep (dat, 2, center_, "-")) ** 2) min_dist <- pmin (min_dist, new_dist) center_index <- c (center_index, new_center) } cur_center <- list () for (i in seq_len (NUM_CLUSTER)) cur_center[[i]] <- unlist (dat[center_index[i],]) dist <- sapply (seq_len (NUM_CLUSTER), function (k) { center_ <- cur_center[[k]] rowSums ((sweep (dat, 2, center_, "-")) **2) }) cur_clusters <- max.col (-dist) return (list (cur_center = cur_center, cur_clusters = cur_clusters)) }, updateCenter = function (dat, cur_clusters) { cur_center <- cbind (dat, cc = cur_clusters) %>% group_by (cc) %>% do (rtn = colMeans (dplyr::select (., -cc))) %>% as.list () %>% "$"(rtn) return (cur_center) }, simple_kmeans = function (dat_, K_ = 2, kmeanspp = TRUE) { dat <- na.omit (dat_[, sapply (dat_, is.numeric)]) NUM_CLUSTER <- as.integer (K_) SIZE <- as.integer (nrow (dat)) if (kmeanspp) tmp <- .self$initCluster_kmeanspp (dat, NUM_CLUSTER) else tmp <- .self$initCluster_random (dat, NUM_CLUSTER) cur_clusters <- tmp$cur_clusters cur_center <- tmp$cur_center old_clusters <- cur_clusters convergence <- FALSE iter <- 1L while (!convergence) { iter <- iter + 1L if (iter > 100L) { cat ("iter reached MAX_ITER\n") break } distances <- sapply (seq_len (NUM_CLUSTER), function (k) { center_ <- cur_center[[k]] d_ <- sweep (dat, 2, center_, "-") return (rowSums (d_ * d_)) }) cur_clusters <- max.col (- distances) print (cur_clusters) if (length (unique (cur_clusters)) != NUM_CLUSTER) if (kmeanspp) cur_clusters <- .self$initCluster_kmeanspp (dat, NUM_CLUSTER)$cur_clusters else cur_clusters <- .self$initCluster_random (NUM_CLUSTER, SIZE)$cur_clusters convergence <- identical (old_clusters, cur_clusters) old_clusters <- cur_clusters cur_center <- .self$updateCenter (dat, cur_clusters) } return (as.integer (cur_clusters)) } ) )
# *********************************************************** # xmeans_sub.r # *********************************************************** xmeans_sub <- setRefClass ( Class = "xmeans_sub", methods = list ( addPackages = function (CRAN = "http://cran.ism.ac.jp/", more = NULL) { addPackages <- c ("dplyr", "matrixStats", "mvtnorm", more) toInstPacks <- setdiff (addPackages, .packages(all.available = TRUE)) for (pkg in toInstPacks) install.packages (pkg, repos = CRAN) for (pkg in addPackages) suppressPackageStartupMessages (library (pkg, character.only = TRUE)) }, xBIC = function (dat_, ignore.covar = TRUE) { dat <- dat_[, sapply (dat_, is.numeric)] nc <- ncol (dat) nr <- nrow (dat) if (ignore.covar) { covar <- dat %>% summarise_each (funs (var)) %>% diag () df <- nc * 2 } else { covar <- cov (dat) df <- nc * (nc + 3) * .5 } center <- colMeans (dat) bic <- -2 * sum (dmvnorm (dat, center, covar, log = TRUE)) + df * log (nr) return (bic) }, xBICp = function (dat_, cluster_, ignore.covar = TRUE) { dat <- dat_[, sapply (dat_, is.numeric)] cluster <- unlist (cluster_) nc <- ncol (dat) nr <- nrow (dat) if (ignore.covar) { covar_eachCluster <- cbind (dat, cc = cluster) %>% group_by (cc) %>% summarise_each (funs (var)) %>% dplyr::select (., -cc) %>% rowwise () %>% do (rtn = diag (.)) %>% as.list () %>% "$"(rtn) sumDeterminant <- sum (sapply (covar_eachCluster, function (mat) {prod (diag (mat))})) df <- 4 * nc } else { covar_eachCluster <- cbind (dat, cc = cluster) %>% group_by (cc) %>% do (rtn = cov (dplyr::select (., -cc))) %>% as.list () %>% "$"(rtn) sumDeterminant <- sum (sapply (covar_eachCluster, function (mat) {det (mat)})) df <- nc * (nc + 3) } center_eachCluster <- cbind (dat, cc = cluster) %>% group_by (cc) %>% do (rtn = colMeans (dplyr::select (., -cc))) %>% as.list () %>% "$"(rtn) beta <- sqrt (c (crossprod (center_eachCluster [[1]] - center_eachCluster [[2]])) / sumDeterminant) logAlpha <- - log (2) - pnorm (beta, log.p = TRUE) logLikelihood_eachCluster <- sapply (seq_along (unique (cluster)), function (k) { center_ <- center_eachCluster[[k]] covar_ <- covar_eachCluster[[k]] dat_ <- dat[cluster == k, ] sum (dmvnorm (dat_, center_, covar_, log = TRUE)) }) bicp <- - 2 * (nr * logAlpha + sum (logLikelihood_eachCluster)) + df * log (nr) return (bicp) }, split2Cluster = function (dat_, ignore.covar = TRUE) { skm <- s_kmeans$new () cluster <- skm$simple_kmeans (dat_, 2) ccounts <- table (cluster) if (any (ccounts < 3)) { return (list (doSPLIT = FALSE)) } bicp <- .self$xBICp (dat_, cluster = cluster) bic <- .self$xBIC (dat_) if (bic > bicp) { return (list (doSPLIT = TRUE, d1 = dat_[cluster == 1, ], d2 = dat_[cluster == 2, ])) } else { return (list (doSPLIT = FALSE)) } }, merge2Cluster = function (dat_i, dat_j) { par <- data.frame (len = c (nrow (dat_i), nrow (dat_j)), val = 1:2) cluster <- par %>% rowwise () %>% do (rval = rep (.$val, each = .$len)) %>% "$"(rval) %>% unlist () dat_ij <- rbind (dat_i, dat_j) bicp <- .self$xBICp (dat_ij, cluster) bic <- .self$xBIC (dat_ij) if (bic < bicp) return (list (doMERGE = TRUE, D = dat_ij)) else return (list (doMERGE = FALSE)) } ) )
# *********************************************************** # xmeans.r # *********************************************************** xmeans <- setRefClass ( Class = "xmeans", contains = c ("s_kmeans", "xmeans_sub"), fields = c ("num_split_cluster" = "integer", "stack" = "list", "ignore.covar" = "logical", "ISHIOKA" = "logical", "Cluster" = "integer"), methods = list ( initialize = function (ignore.covar_ = TRUE, ISHIOKA_ = TRUE) { addPackages () initFields (num_split_cluster = 0L, stack = list (), ignore.covar = ignore.covar_, ISHIOKA = ISHIOKA_) }, ishioka_xmeans = function (DATA_) { .self$splitData (.self$cleanData (DATA_)) if (ISHIOKA) .self$mergeSplitedData () .self$assignCluster () }, splitData = function (data) { # simple x-means split_data <- split2Cluster (data, ignore.covar) if (split_data$doSPLIT) { splitData (split_data$d1) splitData (split_data$d2) } else { num_split_cluster <<- num_split_cluster + 1L stack[[num_split_cluster]] <<- data } }, mergeSplitedData = function () { ind_order <- .self$getDataSize_order (.self$getDataSize_eachCluster ()) num_cluster <- length (ind_order) isMerge_eachCluster <- numeric (num_cluster) for (i in seq_len (num_cluster - 1)) { i_ <- ind_order[sprintf ("%d", i)] if (isMerge_eachCluster[i_] == 1) next for (j in (i+1):num_cluster) { j_ <- ind_order[sprintf ("%d", j)] if (isMerge_eachCluster[j_] == 0) { merge_data <- merge2Cluster (stack[[i_]], stack[[j_]]) if (merge_data$doMERGE) { stack[[i_]] <<- merge_data$D isMerge_eachCluster[j_] <- 1 break } } } } stack <<- stack [isMerge_eachCluster == 0] num_split_cluster <<- length (stack) }, cleanData = function (data_) { out <- na.omit (data_ [, sapply (data_, is.numeric)]) rownames (out) <- 1:nrow (out) return (out) }, getDataSize_eachCluster = function () { out <- sapply (stack, nrow) return (out) }, getDataSize_order = function (DataSize_eachCluster) { out <- seq_len (length (DataSize_eachCluster)) out <- setNames (out, order (DataSize_eachCluster)) return (out) }, assignCluster = function () { out <- numeric (sum (.self$getDataSize_eachCluster ())) ind <- lapply (stack, function (d) as.integer (rownames(d))) for (i in seq_along (ind)) out[ind[[i]]] <- i Cluster <<- as.integer (out) } ) )
# test.r source ("s-kmeans.r") source ("xmeans_sub.r") source ("xmeans.r") xkm <- xmeans$new () xkm$ishioka_xmeans (iris[1:4]) plot (iris[3:4], col = xkm$Cluster, pch = 19)
次のグラフはirisをデータとした場合の結果です。
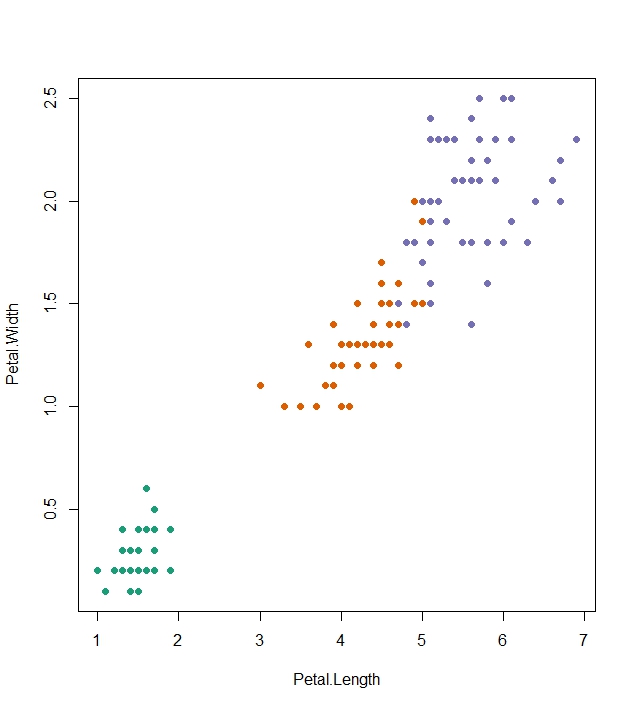
ちなみに、再帰はC言語を学ぶなかで学習しました。C言語は仕事で使うかと思って4月から勉強したのですがいまだ使ってません。しかし、C言語を学ぶとアルゴリズム本が読めるようになるので結果良かったです。
以上です。誰かと未来の自分に参考になれば幸いです。